|

|

|

|
| -- New : transp. journée d'évaluation COST (17/12/04) -- | -- L'agenda -- | -- L'agenda (accès restreint) -- | -- Références -- | -- Homepage Nicolas Dey -- |
En biologie, comme dans pratiquement toutes les disciplines en sciences, les approches scientifiques sont à la fois poussées et limitées par les avancées technologiques. La mise au point de nouveaux outils a donc un impact direct sur les champs d'investigation et les nouveaux concepts qui s'ouvrent aux chercheurs. Aujourd'hui, parallèlement aux techniques et aux concepts de la génomique à haut débit, qui est l'un des moteurs de la recherche en biologie, les avancées techniques en imagerie microscopique sont en train de modifier les approches de la biologie du vivant et de la physiopathologie. La microscopie dynamique à haute résolution permet de visualiser la cellule tant au niveau moléculaire qu'au niveau d'un organisme entier. Elle fournit des informations essentielles à la compréhension de l'organisation spatio-temporelle des processus dynamiques qui structurent les fonctions cellulaires [Galy 00] ainsi que les mécanismes moléculaires des maladies [Voigt 99]. De plus, elle permet d'intégrer un grand nombre des informations obtenues in vitro dans le contexte plus global de processus physiologiques de cellules vivantes. De même, l'imagerie à haute sensibilité permet de sonder les processus moléculaires des interactions des protéines dans les différents compartiments sub-cellulaires et d'acquérir une vue détaillée de la physiologie cellulaire. Ces possibilités sont largement basées sur l'utilisation, d'une part, de molécules fluorescentes qui permettent l'analyse in vivo de la distribution de compartiments cellulaires spécifiques, et, d'autre part, de techniques de microscopie dont les capacités de fonctionnement ont été nettement augmentées par des méthodes numériques de traitement et analyse d'image [Olivo-Marin 02, Zimmer 02].
Pour être en mesure de réaliser ce type d'imagerie à la cadence nécessaire pour suivre des mouvements très rapides, tout en conservant une sensibilité suffisante, la meilleure solution consiste actuellement à réaliser l'imagerie tridimensionnelle de l'échantillon en microscopie à coupes optiques en grand champ et à déconvoluer ensuite le ``stack'' d'images obtenues. Dans ce type de microscope, l'illumination et l'acquisition sont assurées respectivement par un monochromateur à changement de raie ultrarapide et par une caméra CCD refroidie. Un moteur pas à pas guidé par un élément piézoélectrique permet un déplacement calibré suivant l'axe z. En prenant une image à chaque pas en z, on obtient une série d'images parcourant le volume de l'échantillon (``z-stack''). Grâce à tous ces éléments réunis, il est possible d'obtenir des cadences de l'ordre de 80 images/seconde. Par rapport à la microscopie confocale, les images peuvent être obtenues à une cadence très supérieure et avec un meilleur rapport signal/bruit. Cependant, pour obtenir une image tridimensionnelle exacte à partir des coupes optiques, il est nécessaire de déconvoluer le ``stack'' d'images de façon à éliminer la fluorescence non spécifique à chaque plan et à réduire le bruit. De plus, si on utilise des PSF (``Point Spread Function'') spécifiques aux échantillons, la déconvolution permet de restaurer la résolution des images acquises et d'approcher la résolution qui serait obtenue en l'absence d'aberrations et de distorsions. La qualité et la rapidité des algorithmes de déconvolution sont donc des facteurs cruciaux pour l'utilisation de cette méthode d'imagerie.
Le problème de la déconvolution d'images
est inhérent à toute acquisition d'images réelles, qui sont
dégradées par l'optique et
l'électronique utilisées pour leur acquisition. Ce problème
est de modélisation simple : 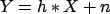 , où
, où 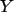 est l'image acquise,
est l'image acquise,
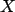 est l'image idéale que l'on cherche, qui est convoluée
avec la fonction de floue
est l'image idéale que l'on cherche, qui est convoluée
avec la fonction de floue 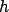 ou PSF et
ou PSF et 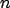 est un bruit additif supposé blanc et gaussien. Nous considérons
le problème en deux temps : d'abord le problème de l'estimation
des dégradations (fonction de flou
est un bruit additif supposé blanc et gaussien. Nous considérons
le problème en deux temps : d'abord le problème de l'estimation
des dégradations (fonction de flou 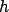 et statistiques du bruit
et statistiques du bruit 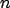 ),
puis la déconvolution (trouver
),
puis la déconvolution (trouver 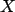 , connaissant
, connaissant 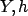 et les
statistiques du bruit
et les
statistiques du bruit 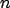 ). Les études antérieures
menées depuis plusieurs années dans le projet Ariana en imagerie
satellitaire ont montré qu'il était nécessaire de traiter ces deux
estimations séparément, les modèles d'images (a priori injectés)
les plus efficaces pour chacun des deux problèmes étant de nature
différente. Pour l'estimation de la fonction de flou, il faut utiliser
un modèle robuste d'images, générique à toutes les images,
utilisant donc des caractéristiques universelles des images comme
l'autosimilarité à travers les échelles. Pour la restauration,
il faut au contraire un modèle plus fin, adapté aux détails de
l'image considérée (contours, textures). Il n'est possible d'estimer les
paramètres d'un tel modèle à partir de l'image floue et
bruitée que si l'on connait les dégradations au préalable.
). Les études antérieures
menées depuis plusieurs années dans le projet Ariana en imagerie
satellitaire ont montré qu'il était nécessaire de traiter ces deux
estimations séparément, les modèles d'images (a priori injectés)
les plus efficaces pour chacun des deux problèmes étant de nature
différente. Pour l'estimation de la fonction de flou, il faut utiliser
un modèle robuste d'images, générique à toutes les images,
utilisant donc des caractéristiques universelles des images comme
l'autosimilarité à travers les échelles. Pour la restauration,
il faut au contraire un modèle plus fin, adapté aux détails de
l'image considérée (contours, textures). Il n'est possible d'estimer les
paramètres d'un tel modèle à partir de l'image floue et
bruitée que si l'on connait les dégradations au préalable.
Concernant la restauration d'images connaissant les dégradations,
il s'agit d'estimer 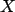 connaissant
connaissant 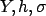 où
où  est
l'écart type du bruit
est
l'écart type du bruit 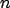 .
La reconstruction d'une image à partir d'une observation floue et
bruitée (cf. fig.2) est un problème mal
posé, quel que soit le type d'acquisition (imagerie satellitaire
ou biologique). Une simple inversion entraîne
une amplification inacceptable du bruit. C'est pourquoi le problème
est souvent régularisé lors de l'inversion, en imposant une contrainte
de lissage à la solution recherchée.
.
La reconstruction d'une image à partir d'une observation floue et
bruitée (cf. fig.2) est un problème mal
posé, quel que soit le type d'acquisition (imagerie satellitaire
ou biologique). Une simple inversion entraîne
une amplification inacceptable du bruit. C'est pourquoi le problème
est souvent régularisé lors de l'inversion, en imposant une contrainte
de lissage à la solution recherchée.
|
Des travaux effectués dans le projet Ariana ont porté sur une nouvelle
approche, appelée COWPATH [Jalobeanu 02]. Le principe de cette
méthode a été
introduit pour la déconvolution d'images par Kalifa et Mallat
[Kalifa 98, Kalifa 99], et consiste à effectuer la
déconvolution sans régularisation dans l'espace
des fréquences, puis à seuiller les coefficients
de la transformée en paquets d'ondelettes de cette image afin
d'éliminer une grande partie du
bruit déconvolué.
Les deux principales difficultés de cette méthode sont la non
invariance par translation due à la transformée en ondelettes réelles
(décimées) et l'estimation des seuils optimaux dans les
sous-bandes. Dans la méthode COWPATH, nous avons proposé d'utiliser
une transformée en ondelettes complexes (et non réelles), introduite
par N. Kingsbury [Kingsbury 98, Kingsbury 99], qui a deux avantages : garantir
l'invariance par translation
de la décomposition donc du résultat du seuillage et offrir une
directionnalité des coefficients en ondelettes. Contrairement à la
décomposition dyadique avec des
ondelettes réelles qui privilégie les directions horizontale et
verticale, la décomposition avec des ondelettes complexes divise le
plan des fréquences en sous-bandes sélectionnant chacune une direction
différente.
La méthode de seuillage utilisée dans l'algorithme COWPATH
[Jalobeanu 02] repose sur
un modèle bayésien de la transformée en ondelettes, qui consiste
à atténuer plus ou moins chaque coefficient en fonction de sa valeur
et de la variance du bruit. Cette méthode permet de régulariser
l'image déconvoluée tout en préservant les contours et les
textures présents dans l'image. De plus, elle est rapide car non
itérative.
Le second problème est celui de l'estimation des dégradations
(ici 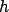 et
et  ), connaissant uniquement l'image observée
), connaissant uniquement l'image observée
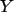 . Si le problème de la déconvolution d'images (trouver
. Si le problème de la déconvolution d'images (trouver 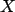 connaissant
connaissant 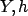 et
et  ) est un problème
mal posé qu'il faut régulariser, celui de la déconvolution aveugle
(trouver en même temps
) est un problème
mal posé qu'il faut régulariser, celui de la déconvolution aveugle
(trouver en même temps 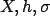 connaissant
connaissant 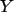 ) l'est
encore plus car le nombre d'inconnues est augmenté sans que le nombre
de données ne change.
A priori, il est
difficile de discerner à partir d'une image floue, quelle est la part
du flou de celle du signal. Plusieurs solutions
peuvent répondre à ce problème, comme par exemple la solution
triviale
) l'est
encore plus car le nombre d'inconnues est augmenté sans que le nombre
de données ne change.
A priori, il est
difficile de discerner à partir d'une image floue, quelle est la part
du flou de celle du signal. Plusieurs solutions
peuvent répondre à ce problème, comme par exemple la solution
triviale 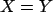 et
et 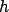 un dirac. Afin de diminuer
l'ensemble des solutions possibles et de régulariser au mieux ce
problème, des contraintes doivent être introduites, à la
fois sur
un dirac. Afin de diminuer
l'ensemble des solutions possibles et de régulariser au mieux ce
problème, des contraintes doivent être introduites, à la
fois sur 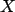 comme cela est fait en déconvolution classique, mais aussi sur
comme cela est fait en déconvolution classique, mais aussi sur
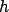 .
.
La plupart des méthodes proposées dans la littérature sur le
sujet utilisent des contraintes fortes sur l'image et sur la
fonction de flou, comme par exemple des contraintes de positivité
(des valeurs négatives ne peuvent correspondre à des signaux
physiques).
Dans les approches explorées dans la littérature, des modèles sont
injectés sur 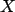 (linéaires,
AR par exemple, ou non linéaires), ainsi que sur
(linéaires,
AR par exemple, ou non linéaires), ainsi que sur 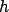 (positivité,
normalisation, décroissance à partir de l'origine...). Ces
méthodes ne conduisent pas à des résultats satisfaisants dans la
mesure où les contraintes injectées ne suffisant pas à régulariser
suffisamment le
problème, les solutions restent multiples. C'est en particulier la
conclusion que nous avons obtenue dans une étude précédente
réalisée pour le CNES
en imagerie satellitaire haute résolution [Jalobeanu 00].
(positivité,
normalisation, décroissance à partir de l'origine...). Ces
méthodes ne conduisent pas à des résultats satisfaisants dans la
mesure où les contraintes injectées ne suffisant pas à régulariser
suffisamment le
problème, les solutions restent multiples. C'est en particulier la
conclusion que nous avons obtenue dans une étude précédente
réalisée pour le CNES
en imagerie satellitaire haute résolution [Jalobeanu 00].
L'algorithme proposé par le projet Ariana, appelé BLINDE
[Jalobeanu 01b, Jalobeanu 01a], pour
estimer la dégradation à partir d'une image
observée (i.e. dégradée), consiste à modéliser
la dégradation dans l'espace de Fourier (FTM) et modéliser la scène
par un mouvement Brownien fractionnaire, dont une caractéristique
importante est la décroissance de la puissance spectrale en loi de
puissance 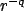 où
où  est la fréquence spatiale radiale. Ce modèle
n'est pas un ``bon'' modèle d'image dans l'absolu, dans le sens où il
ne prend pas en
compte la non-stationarité des images. Cependant, il
traduit la propriété d'invariance par changement d'échelle, qui est
une caractéristique importante et générique des images
naturelles. D'après nos
premiers résultats de test, les images de zones urbaines vérifient aussi
cette propriété. Son adéquation aux images biologiques reste, par contre, à vérifier. La prise en compte de cette propriété semble suffire
pour
discerner le flou et de la scène naturelle, à partir d'une
observation.
est la fréquence spatiale radiale. Ce modèle
n'est pas un ``bon'' modèle d'image dans l'absolu, dans le sens où il
ne prend pas en
compte la non-stationarité des images. Cependant, il
traduit la propriété d'invariance par changement d'échelle, qui est
une caractéristique importante et générique des images
naturelles. D'après nos
premiers résultats de test, les images de zones urbaines vérifient aussi
cette propriété. Son adéquation aux images biologiques reste, par contre, à vérifier. La prise en compte de cette propriété semble suffire
pour
discerner le flou et de la scène naturelle, à partir d'une
observation.
L'estimation s'effectue au sens du maximum de
vraisemblance marginalisée, approché dans l'algorithme proposé.
Les résultats obtenus par cette méthode sont très bons. La qualité
obtenue est supérieure à celle obtenue avec d'autres méthodes
proposées dans la littérature, dont certaines ont été explorées dans
l'étude précédente [Jalobeanu 00]. De plus, on peut noter que
l'algorithme d'estimation
est rapide. Cet algorithme a fait l'objet d'un dépôt de brevet
[Jalobeanu 01c] et a été testé avec succès par le CNES lors
de la recette en vol du satellite SPOT5 lancé en mai 2002 (cf.
paragraphe 2.4).
Concernant la partie estimation de la fonction de flou
(déconvolution aveugle), le travail théorique
pour faire évoluer BLINDE vers le problème de l'imagerie biologique
est plus important. Il s'agit d'abord de proposer et tester un modèle
mathématique paramétré de la fonction de flou, ainsi qu'un
modèle de bruit, pour modéliser entièrement les
dégradations dues au microscope. Ce modèle doit être le plus
possible fidèle à la physique du système optique d'imagerie afin d'être le plus
précis possible. Il faut ensuite, et il s'agit là d'une
grande partie du travail, proposer une modélisation robuste et
générique des images biologiques, constituant un a priori sur les
images biologiques pour l'estimation de la fonction de flou. Cet a
priori sera, en effet, différent de celui utilisé pour des
images d'observation de la Terre.
Le test des algorithmes sera effectué pendant la phase de
développement sur des images biologiques de référence issues de
la recherche en biologie cellulaire.
Cette partie du projet permettra de valider les hypothèses sur la
modélisation du système optique d'imagerie et le modèle de
bruit. Il permettra également d'étudier l'influence des
différents paramètres instrumentaux (grossissement, longueur
d'onde, exposition, ouverture) sur la qualité de la
déconvolution. Une attention particulière sera portée aux
dégradations introduites à la fois par la non planéité des
échantillons et par la variation d'index du milieu cellulaire avec
la profondeur. Une dernière phase de test du projet consistera à
comparer les résultats obtenus sur les images tests à ceux
fournis par les logiciels commerciaux de référence, tant en
termes de qualité de restauration que de rapidité. A l'issue de
ces tests, le logiciel sera mis à la disposition de chercheurs en
biologie pour une validation en condition de routine.
Participants : Josiane Zerubia (DR1 INRIA), Laure Blanc-Féraud (DR2 CNRS), Nicolas Dey (Ph.D.).
Le rôle du projet Ariana sera d'apporter son expertise sur les
méthodes de déconvolution aveugle pour l'analyse d'images, dans
le cadre des applications spécifiques intéressant l'Institut
Pasteur et l'Institut Weizmann. Il s'agira d'étendre au 3D et à
l'imagerie biologique une méthode de déconvolution aveugle
(logiciels COWPATH2 et BLINDE, dépôts à l'APP (Agence pour la
Protection des Programmes) et brevet en juillet 2001
[Jalobeanu 01c]) initialement dédiée à l'imagerie
satellitaire. Ce travail demande un réel effort de recherche
théorique car les problèmes posés en biologie et les modèles
liés aux capteurs sont différents.
(I.N.R.I.A. - Projet ARIANA)
Participants : Jean-Christophe Olivo-Marin (DR2 IP, Unité d'Analyse d'Images Quantitative (AIQ) ,CNRS URA 1947), Nancy Guillen (DR2 CNRS, Unité Biologie Cellulaire du Parasitisme (BCP), INSERM U 389).
Le rôle de l'Unité AIQ sera d'apporter son expertise en microscopie et en
traitement et analyse d'images microscopiques en biologie. Il s'agira
de bien intégrer les spécificités de l'imagerie biologique
dans les modèles mathématiques et de tenir compte des
difficultés particulières liées à l'utilisation de matériel
vivant pour la reproductibilié et la vérification des mesures.
L'Unité AIQ aura aussi un rôle d'interface avec les équipes de
biologie, en particulier l'unité BCP.
(Institut Pasteur)
Participants : Benjamin Geiger (Professeur, IW), Zicki Kam (Professeur, IW).
Le rôle de l'Unité MCB sera d'apporter son expertise en
microscopie et en biologie cellulaire pour la validation de la
méthode de déconvolution sur des images issues de projets de
recherche en biologie. Cette expertise, qui comprend aussi une longue
pratique de la déconvolution en microscopie, sera précieuse pour
juger de l'intégrité des données après traitement ainsi que
de la relevance biologique des résultats.
(Weizmann Institute of Science)
Coordinateur de la proposition ARC: Josiane Zerubia, I.N.R.I.A..
| -- New : transp. journée d'évaluation COST (17/12/04) -- | -- L'agenda -- | -- L'agenda (accès restreint) -- | -- Références -- | -- Homepage Nicolas Dey -- |