MODE : Myopic Optical DEconvolution
Action COLOR 2009 de l’INRIA Sophia Antipolis Méditerranée
Objet de la proposition
Le but de cette action COLOR est de proposer un nouveau protocole d’acquisition et de nouvelles méthodes d’estimation permettant d’améliorer la qualité des images biologiques acquises par microscopie confocale, dans le cas de certains marqueurs fluorescents. Il s’agit de marqueurs fluorescents émettant dans une large bande spectrale comme le DAPI, un intercalant fluorescent de l’ADN très largement utilisé pour la visualisation spécifique des noyaux cellulaires et des chromosomes de cellules animales et végétales, ainsi que pour identifier et quantifier l’ADN (l’ensemble des gènes) dans ces organelles.
Nous proposons d’acquérir conjointement deux images du même objet, à deux longueurs d’ondes différentes. Nous voulons montrer dans cette étude que l’on peut utiliser ces informations pour améliorer la restauration des images, en particulier améliorer la résolution en profondeur. Le but de cette action COLOR est :
- d’acquérir des images biologiques dans les conditions particulières prédéfinies pour l’étude,
- développer et programmer une méthode de restauration d’images pour ce type de données,
- appliquer cette méthode sur des spécimens en biologie animale et végétale et montrer l’intérêt de cette démarche.
Equipes concernées
- EPI ARIANA, Laboratoire I3S (UNSA /CNRS) et INRIA Sophia Antipolis Méditerranée
- Institut de Biologie du Développement et Cancer de Valrose, UMR CNRS/UNSA
- INRA Sophia Antipolis
Descriptif scientifique (3 pages maximum).
L’imagerie biologique a fait de considérables avancées ces dernières années, avec l’apparition sur le marché de nombreux systèmes d’imagerie. Parmi ceux-ci, la microscopie confocale par fluorescence offre la possibilité d’imager des spécimens vivants en 3D à une résolution de l’ordre de quelques centaines de nanomètres. Malgré le système confocal, l’image présente un flou, en particulier en profondeur. Il est possible d’améliorer la résolution des images par déconvolution. En effet, le modèle d’observation de ces images est une convolution entre l’image réelle du spécimen et la fonction d’étalement de l’appareil optique, appelée PSF pour « Point Spread Function ». La restauration de ces images consiste donc en une déconvolution 3D en présence de bruit. L’amplification du bruit lors de la déconvolution peut être atténuée par une régularisation adaptée aux images (voir l’article [N. Dey, L. Blanc-Féraud, C. Zimmer, Z. Kam, P. Roux, J.C. Olivo-Marin et J. Zerubia ''Richardson-Lucy Algorithm with Total Variation Regularization for 3D Confocal Microscope Deconvolution.'' Microscopy Research Technique, 69: pages 260-266, 2006]). Une des difficultés de la restauration est la connaissance précise de la PSF. Les modèles mathématiques de la physique d’acquisition en donnent une expression en fonction des paramètres physiques liés aux conditions expérimentales. Ceux-ci ne sont pas toujours connus avec exactitude ou peuvent varier d’une expérience à l’autre (comme la température ou l’indice optique de l’échantillon). Il s’avère utile de développer des techniques d’estimation de la PSF en même temps que la déconvolution, à partir de la seule image observée. C’est un problème inverse dit « mal posé » car l’ensemble des solutions possibles est grand et les solutions trouvées ne sont pas stables par rapport aux données observées.
Dans le cas de certaines sondes ayant un spectre d’émission large, il est possible d’observer le même objet dans différents canaux. Le cas du DAPI est caractéristique de ce phénomène ; excité à partir d’un laser UV, on observe son signal d’émission entre 400 et 600nm. Il est donc possible d’acquérir simultanément des images sur différentes gammes de longueurs d’onde (par exemple 390-465nm et 565-615nm), générant ainsi des PSF différentes. La réémission du spécimen étant la même autour des deux longueurs d’ondes considérées, nous aurons ainsi à notre disposition deux images observées issues de la convolution du même objet avec deux PSF différentes. Sous certaines conditions, en général vérifiées dans les expériences, et dans le cas d’un bruit additif blanc et gaussien sur les images, il est montré dans [G. Harikumar and Y. Bresler, ``Blind restoration of images blurred by multiple filters: theory and efficient algorithms,'' IEEE Trans. Image Processing, vol. 8, pp. 202--219, Feb. 1999] que les filtres et l’image peuvent être estimés exactement en l’absence de bruit et de manière stable en présence de bruit. Dans le cas de l’imagerie confocale, nous sommes en présence d’un bruit de Poisson. Même si les résultats théoriques précédents ne s’appliquent pas directement, nous serons en mesure de développer une méthode de restauration multibandes adaptées aux données. Cette méthode améliorera sans aucun doute les résultats par rapport à ceux obtenus avec une seule image, ne serait-ce que parce le problème de la déconvolution myope (c’est-à-dire où la PSF est connue à des paramètres près) est alors mieux posé car nous avons réduit le rapport du nombre de variables inconnues sur le nombre de données. De plus, déconvoluer une image à partir de deux observations différentes doit permettre de réduire l’effet du bruit.
Le but de cette action COLOR est donc de développer une nouvelle méthode de restauration d’image 3D en imagerie confocale avec estimation de la fonction de flou, à partir de données acquises dans les conditions prédéfinies. La proposition comprend l’acquisition des images, l’étude théorique du problème de la déconvolution en présence de bruit de Poisson à partir de deux images observées et le développement d’une méthode adaptée, puis la validation de l’approche sur des données réelles biologiques.
Les tests se feront d’abord en imagerie par microscopie confocale sur des spécimens simples de biologie animale, en collaboration avec l’Institut de biologie du Développement et du Cancer, puis sur des spécimens de biologie végétale plus complexes à imager, en collaboration avec l’INRA. Nous espérons améliorer la résolution des images acquises par microscopie confocale. La qualité des résultats attestant de la validité de la méthode proposée sera mesurée par des biologistes.
Le cas du marqueur DAPI que nous proposons d’utiliser pour cette étude est particulier, puisqu’il est spécifique à l’ADN. Cependant, si les résultats sont concluants avec ce marqueur, il sera possible d’envisager d’étendre la méthode à d’autres marqueurs qui, eux, pourraient être utilisés pour de l’immuno-fluorescence, et ainsi permettre de localiser des protéines cibles avec une meilleure résolution. Une autre application possible est en imagerie des végétaux où on utilise la fluorescence naturelle des végétaux. En effet, presque toutes les plantes, en fonction de leur âge, ont, dans les parois de certaines de leurs cellules, des molécules très complexes comme des lignines. Les spectres représentés dans la figure ci-dessous montrent qu’en utilisant le laser 405 nm la lignine produit un spectre à large bande et nous pourrons envisager d’utiliser la méthode proposée dans cette action. Les plantes modèles utilisées couramment en biologie moléculaire et cellulaire sont par exemple le tabac et la petite crucifère, comme la moutarde sauvage, Arabidopsis thaliana. Ces spécimens pourront être fournis par l’INRA.
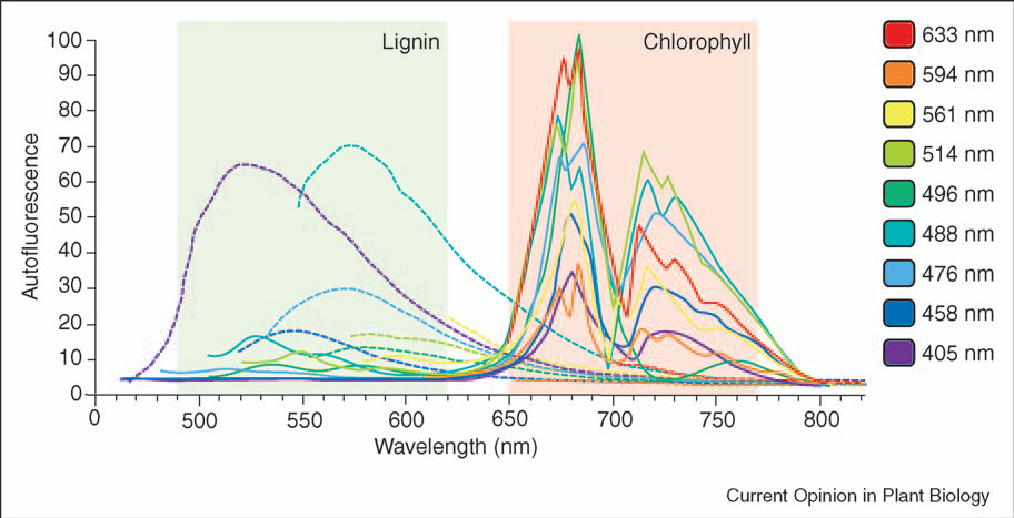
Spectres d’émission des principales sources d’autofluorescence des racines et des feuilles après excitation avec des sources laser standard de microscope confocale, à 9 longueurs d’onde différentes. La lignine (en pointillé) et la chlorophylle (en trait plein) sont utilisées respectivement pour les racines et les feuilles. On voit par exemple que la lignine émet fortement entre 490 et 620 nm et la chlorophylle entre 650 et 770 nm.
Modalités de la collaboration (préciser le nom et la spécialité des personnes impliquées dans chaque équipe),
- EPI ARIANA : Laure Blanc-Féraud, DR CNRS et Josiane Zerubia, DR INRIA. Spécialistes en traitements des images, elles apporteront leur connaissances et compétences dans les modèles et méthodes numériques de restauration des images biologiques.
- IBDC/Plateforme d’imagerie PRISM : Sébastien Schaub, IR CNRS. Chargé du développement technique de la plateforme, il apportera ses compétences en microscopie, et mettra à disposition les différents microscopes confocaux pour les acquisitions.
- INRA Sophia Antipolis : Gilbert Engler, IR INRA. Responsable du plateau de microscopie à l’INRA Sophia Antipolis. Spécialiste en Biologie Cellulaire Végétale avec des compétences spécifiques dans le domaine du cytosquelette et du cycle cellulaire chez les végétaux. Il apportera ses compétences pour la valorisation des méthodes proposées.
Autres actions du projet sur la biologie
L’EPI Ariana travaille sur le sujet de la restauration d’images biologiques acquises par microscopie confocale en collaboration avec Jean-Christophe Olivo-Marin du laboratoire d’Analyse Quantitative d’Image de l’Institut Pasteur à Paris et le professeur Zvi Kam de l’institut Weizmann en Israël dans le cadre d’un P2R initié en 2004. Un second P2R a suivi en 2006 avec la collaboration supplémentaire du professeur Arie Feuer du Technion en Israël. Le sujet est la déconvolution d’images et l’estimation de la fonction de flou à partir d’une seule image de spécimen acquise. Ce travail s’effectue en particulier dans le cadre de la thèse de Praveeen Pankajakshan qui soutiendra fin 2009. Le projet Ariana a donc une forte expertise dans le domaine de la déconvolution aveugle à partir d’une seule image, mais le problème de la restauration à partir de deux images avec deux fonctions de flou différentes n’a jamais été traité. Le sujet de la COLOR est donc complémentaire aux travaux effectués dans le projet Ariana, au cours des programmes mentionnés.
L’EPI Ariana travaille actuellement pour proposer un projet d’ANR dans le programme DEFIS en 2009, sur le sujet de la déconvolution en imagerie biologique avec d’autres partenaires, incluant l’INRA de Sophia Antipolis, mais pas le laboratoire de biochimie de Valrose. Le sujet est la déconvolution d’images utilisant des informations supplémentaires, différentes de celles proposées ici. Il s’agit d’utiliser l’information temporelle dans le cas de séquence d’images ou une information donnée par un autre type de modalité, par exemple la tomographie. Là encore, il y aura complémentarité si l’ANR est acceptée en 2009.
Revenir au site de l'INRIA.