Segmentation automatique : application aux angioscanners 3D du foie
Luc SOLER
Grégoire MALANDAIN
Hervé DELINGETTE
Version finale (postScript)
Introduction
Dans le cadre de la réalisation d'un simulateur d'opérations
laparoscopiques, nous avons développé une nouvelle méthode permettant
d'extraire dans les angioscanners 3D du foie, le réseau vasculaire de
la veine porte. Ce réseau est utilisé en pratique pour repérer les 8
différents segments anatomiques qui représentent l'unité de base dans
les ablations chirurgicales sur le foie. Notre méthode réalise cette
segmentation en trois étapes réalisant tout d'abord un seuillage
globale suivi d'une correction locale, en terminant par une analyse
topologique et géométrique. Elle permet ainsi de segmenter précisément
ce réseau difficile à délimiter dans la pratique et ainsi de
reconstruire les 8 segments anatomiques à partir d'une image acquise
par un protocole standard.
État de l'art
D'un point de vue général, la segmentation est aujourd'hui un problème
très étudié en analyse d'images médicales [Aya95]. Les méthodes de
segmentation ont été développées suivant deux axes principaux, les
méthodes d'analyse globale de l'image et les méthodes d'analyse
locale. Les premières réalisent une classification par seuillage pour
laquelle les seuils sont calculés à partir de l'histogramme
d'intensité de l'image complète [HFC96,G93,SSW88], suivie généralement
d'opérations morphologiques [FKHG96,BMEL93]. Parmi ces méthodes,
plusieurs ont pour but d'ajuster des fonctions sur l'histogramme des
intensités de façon à ce que la somme de ces fonctions soit une
approximation de cet histogramme [GO94,CHY89,Der87]. Chacune des
fonctions peut alors représenter une classe différente dans
l'image. Dans [FKHG96], l'histogramme est décomposé en trois zones
représentant trois classes de tissus dans le foie : les tumeurs, le
tissu hépatique et les vaisseaux. La délimitation de ces classes est
calculée ici par la méthode de classification par pourcentage [NFM90]
moins précise que les méthodes d'ajustement de fonctions.
Les méthodes d'analyse locale quant à elles, réduisent le traitement
de l'image à des zones plus limitées, ce qui fournit en général un
résultat plus précis, mais plus difficile à automatiser. La
réduction peut consister à découper l'image en sous-parties plus
petites, chacune d'elles étant alors analysée par les méthodes de
seuillage vue précédemment [KS90]. La majorité de ces méthodes reste
cependant les méthodes par croissance de régions qui réalisent une
segmentation locale le long d'une vague de propagation par un simple
seuillage avec un seuil fixe [ZJP95,ZJELPK95] ou variable
[SCHP94,SPH93]. Parfois la méthode de segmentation utilisée le long de
la vague est plus complexe, faisant intervenir un seuillage non plus
sur l'intensité, mais sur le gradient d'intensité [OE94], ou encore
des propriétés de courbure de la vague [Sun89]. Mais l'inconvénient
majeur de ces méthodes est l'évaluation automatique des seuils à
utiliser (sur le gradient ou sur l'intensité), ainsi que la définition
du point de départ de la vague de propagation.
Étude théorique et implémentation
En analysant l'étude précédente, nous avons choisi de tirer partie,
autant que possible, des avantages des méthodes de segmentation
existantes par une segmentation en trois étapes précédée d'une étape
de prétraitement de l'image.
Le prétraitement de l'image a pour but de réduire l'image à la
zone d'intérêt délimitée par les contours du foie. Nous utilisons pour
cela l'algorithme développé par Montagnat et Delingette [MD97] qui
déforme un maillage de référence en utilisant des contraintes de
déformation hybrides, locales et globales. Comme le montre la figure
suivante, ceci a pour effet de limiter l'histogramme des intensités,
ce qui facilitera ainsi les traitements ultérieurs.
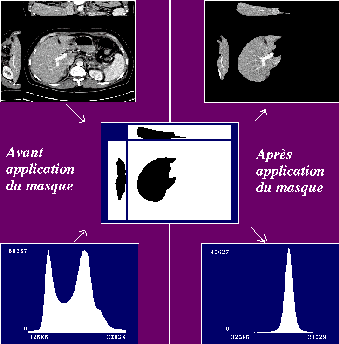
Réduction de l'image au contour du foie et effet sur l'histogramme des intensités.
Le prétraitement étant réalisé, nous souhaitons dans une première
étape segmenter les trois classes décrites dans [FKHG96] (les
lésions, le tissu hépatique et les vaisseaux), en calculant deux seuils S_LF
et S_FV permettant de séparer par simple seuillage ces trois
classes. Pour calculer ces seuils, nous avons supposé que la
distribution des intensités de chacun des tissus suivait une loi
normale. La solution que nous avons choisi est alors de retrouver les
trois distributions ajustant au mieux l'histogramme par la méthode aux
moindres carrées de Levenberg-Marquardt [PFTV91], et d'utiliser
l'intersection de deux distributions voisines comme seuil. Cette
méthode étant très dépendante de l'initialisation, nous avons
développé un ajustement en deux temps. Tous d'abord nous ajustons une
première gaussienne sur le pic principal de l'histogramme qui
correspond au foie et qui est nettement détaché. En utilisant le
résultat de cet ajustement et en le soustrayant à l'histogramme
initial, on peut ainsi repérer deux zones de part et d'autre de ce
pic. En utilisant cette soustraction on peut donc initialiser deux
autres gaussiennes et recommencer l'ajustement avec trois gaussiennes
(voir
figure). On obtient ainsi un premier seuillage automatique mais
imparfait puisque tous les voxels d'un tissu ne seront pas récupérés
et que certains voxels du tissu voisin le seront par erreur (exemple
de seuillage du réseau vasculaire).
La seconde étape a pour but d'améliorer le résultat de la
segmentation des vaisseaux, en récupérant les branches que la première
étape avait mal segmentées. Une solution serait de réduire le seuil S_FV
et de réaliser un seuillage par hystérésis, mais la réduction du seuil
ajouterait alors plus de voxels appartenant aux tissus hépatiques
qu'aux vaisseaux. Afin de limiter ces erreurs, nous avons limité ce
seuillage au voisinage des voxels des vaisseaux issus de la
première segmentation. En effet, plus on sera proche de ces voxels,
plus on aura de chance d'appartenir aux vaisseaux et donc plus on
pourra être permissif. Nous avons ainsi défini un seuil bas variant
en fonction de la distance aux voxels issus de la première
segmentation et illustré sur la figure suivante.
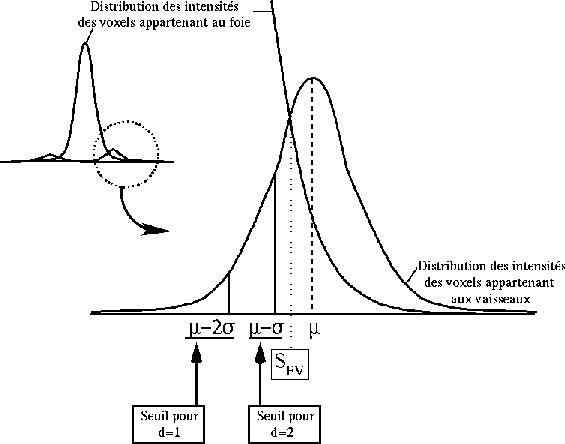
Choix des seuils en fonction de la distance par rapport aux voxels
des vaisseaux.
Compte tenu de l'anisotropie de nos images qui
ont une épaisseur de coupe au moins trois fois plus importante que la
taille du pixel, nous avons fixé une distance maximale d'analyse à
deux pixels, ce qui rend le calcul 3D inutile et accélère donc
l'algorithme. Cette analyse locale nous permet ainsi de récupérer des
branches supplémentaires comme nous pouvons le voir sur l'exemple
suivant.
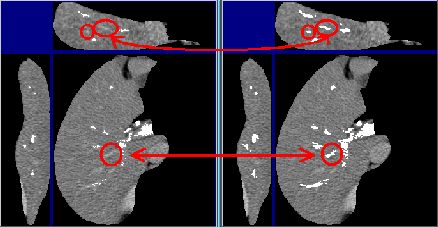
Simple segmentation (à gauche) et apport de l'analyse locale (à droite).
La troisième étape a pour but d'extraire dans les réseaux
vasculaires segmentés, le réseau de la veine porte. Pour cela nous
réalisons une étude topologique et géométrique du résultat de la
segmentation issue de l'étape précédente. La méthode que nous avons
développé est comparable aux méthodes par croissance de
région. Elle réalise en fait une propagation sur l'arbre défini par le
squelette des vaisseaux segmentés. Ainsi, au lieu de réaliser une
propagation sur l'image initiale (voir
figure), elle s'applique uniquement au squelette des vaisseaux
[MBA93] (voir
figure). Comme le montre les figures précédentes, cette méthode
permet de repérer de façon plus précise les points de bifurcation des
branches qui sont couramment utilisés pour définir les plans de coupe
des segments anatomiques. De plus, contrairement aux méthodes
classiques par croissance de région, celle-ci utilise des critères de
sélection basés sur la topologie et la géométrie du réseau traité,
permettant de supprimer les points ou les branches connectées par
erreur. Cette suppression est basée sur l'analyse anatomique du réseau
porte. Ce réseau est un arbre dont chaque branche décroît en diamètre
à chaque bifurcation. En réalisant un parcourt partant du tronc de la
veine porte, on élimine donc les branches qui réalisent une boucle
dans le réseau ou bien qui ont un diamètre qui augmente. Ceci permet
d'éliminer les erreurs de segmentation dues à l'anisotropie des images
(connexion entre réseaux porte et sus-hépatiques, voir figure), mais également de déconnecter le
réseau artériel quant il apparaît (voir
figure).
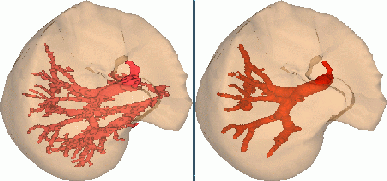
Extraction automatique de la veine porte : Déconnexion du réseau
sus-hépatique.

Extraction automatique de la veine porte : Déconnexion de l'artère hépatique.
Expérimentation
Nous avons testé notre algorithme sur un ensemble de 10 images acquises
par scanner X suivant des protocoles différents. L'analyse progressive
des résultats obtenus à partir des différentes modalités a permis de
définir un protocole d'acquisition standard pour les images du foie
obtenues par scanner X. Ce protocole est ainsi défini par une
injection du produit opaque au rayon X par intraveineuse, suivie d'une
acquisition scanner au temps portal de 50s (veine porte contrastée)
avec une épaisseur de coupe optimale de 2 mm (et maximale de 3 mm) à
la reconstruction pour une taille de pixel approximative de 0,6
mm. Les résultats obtenus sur dix patients et vérifiés par un
radiologue, montrent que l'algorithme extrait automatiquement les
trois principales bifurcations de la veine porte, aussi bien qu'une
segmentation manuelle. Cela permet ainsi de délimiter les segments
anatomiques définis dans l'anatomie conventionnelle du foie. Nous
avons donc créé ces segments automatiquement à partir de l'étiquetage
manuel des vaisseaux segmentés par notre algorithme. Les premiers
résultats obtenus sont très encourageant et montrent un faible écart
entre le modèle anatomique et le modèle issu de l'image, comme le
montre la figure suivante.
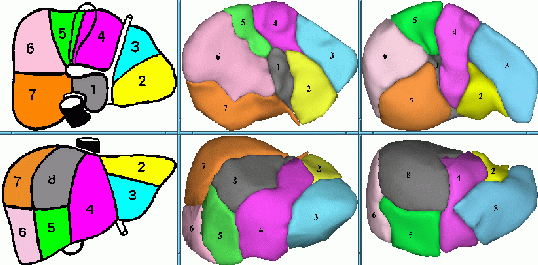
Délimitation des segments anatomiques suivant le modèle de Couinaud.
Application
Nos travaux ont été développé dans le cadre du projet Européen
"Master", réalisé en collaboration avec l'IRCAD (Institut de Recherche
en Cancérologie de l'Appareil Digestif, Hôpitaux de Strasbourg, 1
place de l'Hôpital, 67091 STRASBOURG). Le but est d'apporter aux
chirurgiens une aide essentielle lors de la phase préopératoire, en
facilitant l'analyse des images et la planification des interventions
chirurgicales, mais également en optimisant le geste chirurgical.
En effet, dans la pratique, les chirurgiens utilisent la définition de
Couinaud [coui59], qui permet de repérer les segments à partir de
repères issus essentiellement de la veine porte (voir
figure). Ils délimitent ainsi les 8 segments manuellement à partir
d'images souvent difficiles à analyser. L'anisotropie de ces images
rend souvent plus complexe cette analyse, le suivie mental des
arborescences interne étant alors une tâche d'autant plus difficile et
subjective.
L'automatisation de ce traitement qu'apporte notre algorithme est donc
une aide essentielle aux chirurgiens. Les interactions
avec le Pr. Marescaux, le Dr. Russier et le Dr. Tassetti, chirurgiens
à l'IRCAD, ont permis la validation des résultats que nous avons
obtenus, en particulier dans le domaine de l'étiquetage suivant le
modèle anatomique de Couinaud. Le Pr. Roy, radiologue à l'IRCAD, nous
a fourni pour sa part, l'ensemble des images sur lesquels nous avons
testé notre algorithme, permettant ainsi l'élaboration d'un protocole
d'acquisition plus efficace.
Remerciements
Nous remercions l'IRCAD Strasbourg et plus particulièrement Jean-Marie
Clément ingénieur de recherche, le Pr. Marescaux, le Dr. Russier, le
Dr. Tassetti et le Pr. Roy. Nous souhaitons associer à ces remerciements
toute l'équipe EPIDAURE de l'INRIA Sophia Antipolis au sein de
laquelle ces travaux ont été réalisés et plus particulièrement son
responsable Nicholas Ayache.
Références
- [Aya95] N. Ayache. "Medical computer vision,
Virtual Reality and Robotics". Image and Vision
Computing, 13(4):295-313, mai 1995.
- [BMEL93] M.E. Brummer, R.M. Mersereau,
R.L. Eisner, et R.R.J. Lewine. "Automatic Detection of Brain Contours
in MRI Data Sets". IEEE Trans. on Medical Imaging,
12(2):153-166, juin 1993.
- [CHY89] S. Cho, R. Haralick, et
S. Yi. "Improvement of Kittler and Illingworth's Minimum Error
Thresholding". Pattern Recognition, 22(5):609-617, 1989.
- [Der87] H. Derin. "Estimating Components of
Univariate Gaussian Mixtures Using Priony's Method". IEEE Trans. on
Pattern Analysis and Machine Intelligence, PAMI-9(1):142-148,
janvier 1987.
- [FKHG96] E.K. Fishman, B.S. Kuszyk,
D.G. Heath, et L. Gao. "Surgical Planning for Liver
Resection". IEEE Computer, 29(1):64-72, janvier 1996.
- [G93] C.A. Glasbey. "An Analysis of
Histogram-Based Thresholding Algorithms". Graphical Models and
Image Processing, 55(6):532-537, novembre 1993.
- [GO94] A. Goshtasby et
W.D. O'Neill. "Curve Fitting by a sum of Gaussians". CVGIP
Graphical Models and Image Processing 56(4):281-288, juillet 1994.
- [HFC96] L. Hayat, M. Fleury, et
A.F. Clark. "Candidate Functions for a Parallel Multi-level
Thresholdinf Technique". Graphical Models and Image
Processing 58(4):360-381, juillet 1996.
- [KS90] D.P. Kottke et
Y. Sun. "Segmentation of Coronary Arteriograms by Iterative Ternary
Classification". IEEE Trans. on Biomediacl Engineering,
37(8):778-785, août 1990.
- [MBA93] G. Malandain, G. Bertrand, et
N. Ayache. "Topological Segmentation of Discrete
Surfaces". Internationnal Journal of Computer Vision,
10(2):183-197, février 1993.
- [MD97] J. Montagnat et
H. Delingette. "Volumetric Medical Images Segmentation using Shape
Constrained Deformable Models". Computer Vision, Virtual
Reality and Robotics in Medicine, 1997.
- [NFM90] D.R. Ney, E.K. Fishman, et
D. Magid. "Volumetric Rendering of Computed Tomography Data:
Principles and Techniques". IEEE Computer Graphics and
Applications, pages 24-32, mars 1990.
- [OE94] J.F. O'Brien et
N.F. Ezquerra. "Automated Segmentation of Coronary Vessels in
Angiographics Image Sequences Utilizing Temporal, Spatial and
Structural Constraints". Visualization in Biomedical
Computing, Rochester, MN., pages 25-38, 1994.
- [PFTV91] W.H. Press, B.P. Flannery,
S.A. Teukolsky, et W.T. Vetterling. "Numerical Recipes in
C". Cambridge, 1991.
- [SCHP94] A.R. Sanderson, E. Cohen,
T.C. Henderson, et D.L. Parker. "Vascular Models: from Raw Data to
Geometric Models". Spring Symposium Series, Applications of
Computer Vision in Medical Image Processing, Stanford, CA., mars 1994.
- [SPH93] A.R. Sanderson, D.L. Parker, et
T.C. Henderson. "Simultaneous Segmentation of MR and X-ray
Angiograms for Visualization of Cerebral Vascular
Anatomy". Int. Conf. Volume Image Processing, Utrecht, The
Netherlands, juin 1993.
- [SSW88] P.K. Sahoo, S. Soltani, et
A.K.C. Wong. "A Survey of Thresholding Techniques". Computer
Vision, Graphics and Image Processing, 41:233-260, 1988.
- [Sun89] Y. Sun. " Automated Identification
of Vessel Contours in Coronary Arteriograms by an Adaptative Tracking
Algorithm". IEEE Trans. on Medical Imaging, 8(1):78-88,
mars 1989.
- [ZJELPK95] C.Zahlten, H. Jurgens,
C.J.G. Evertsz, R. Leppek, H.O. Peitgen, et K.J. Klose. "Portal
Vein Reconstruction Based on Topology". European Journal of
Radiology, 19:96-100, 1995.
- [ZJP95] C.Zahlten, H. Jurgens,et
H.O. Peitgen. "Reconstruction of branching blood vessels from
CT-data". Visualization in Scientific Computing,
Springer-Verlag, Wien, pages 41-52, 1995.






