Développement d'algorithmes pour un simulateur
d'environnement marin piloté par ordinateurs
SEMPO
Financé par le programme
Bioinformatique
inter EPST
Porteur du projet : O.Bernard
(INRIA Sophia-Antipolis)
Equipes impliquées :
|
.
|
O.Bernard, J.-L.Gouzé, C.Prévost
|
|
|
Y.Leredde, J.-C.Poggiale, D.Nérini, C.Manté,
|
CNRS URA 2077, L.O.V.
(Laboratoire d'Océanographie de Villefranche)
|
L.Pawlowski et
A .Sciandra
|
Objectif du projet :
L'objectif du projet est de reproduire expérimentalement l'environnement
de cellules phytoplanctonique tel qu'il peut être rencontré
en milieu marin. Les expériences réalisées à
l'aide de ce simulateur de milieu marin permettront alors de mieux étudier
le mécanisme d'absorption du carbone par le phytoplancton au cours
de la photosynthèse. Les modèles obtenus devraient donc contribuer
à améliorer les prédictions de la quantité
de carbone fixée durant la production primaire, ils permettront
donc d'affiner l'estimation des flux de carbone entre l'océan et
l'atmosphère.
Résumé des travaux :
Un nouveau modèle décrivant la croissance du phytoplancton
co-limité par l’azote, l’intensité lumineuse et la température
a été développé et validé. Ce modèle
a été couplé au logiciel de simulation hydrodynamique
côtière SIMPHONIE afin de simuler l’évolution 3-D des
distributions spatiales des variables physiques, chimiques et biologiques
au sein de la colonne d’eau. Les champs obtenus ont ensuite été
utilisé pour reconstruire les trajectoires lagrangiennes des particules.
Ce code de simulation a donc permis de recalculer l’environnement physico-chimique
de cellules de phytoplancton, en fonction de leur position initiale. Des
procédures de classement ont alors abouti à 7 classes d’environnements
perçues par les algues. Parallèlement, le logiciel de pilotage
des bio réacteurs, qui permettra de reconstruire expérimentalement
ces trajectoires a été conçu à l’aide d’un
formalisme UML et son développement en langage JAVA est en cours.
Budget (montant total alloué
pour les 2 années: 110 kF = 16.75 kEuros):
Dépenses entre décembre 2000 et novembre 2002:
| Missions
(5 visites) |
Année 1
Année 2 |
|
| PC LOB |
Année 1 |
3048Euros
|
| PC LOV |
Année 1 |
2980Euros
|
| Stages |
Année 1 (O. Croce)
Année 2 (G.Cesari)
Année 2 (G.Mauguin*)
Année 2 (C.Pénard) |
4470Euros
2270 Euros
2354 Euros
1600 Euros
|
| Total |
Année 1
Année 2 |
|
| |
TOTAL |
17 645 Euros
= 115 751 FF |
(*) le stage de G.Mauguin a été
en majeure partie financé par l'action INRIA COLOR "Hydrobio" (5340
Euros)
Résumé du projet:
 Développement
d'un logiciel pour piloter et synchroniser les différents automates
de culture et stocker les données dans une base.
Développement
d'un logiciel pour piloter et synchroniser les différents automates
de culture et stocker les données dans une base.
 Résumé
des travaux: L'objectif est de centraliser le pilotage des différents
automates de culture qui jusqu'à présent ne communiquaient
pas entre eux, de rassembler l'information récoltée et de
la stocker dans une base de données. Enfin, ce logiciel devra permettre
la mise en œuvre des algorithmes de contrôle sur la base des diverses
informations recueillies. La maquette du logiciel a été réalisée,
en TCL/Tk. Les flux d’information et l’interaction entre les divers modules
ont été analysés et modélisés selon
une approche UML. Le logiciel de pilotage est centré sur une base
de données MySQL qui contient les paramètres des divers automates,
et recueille les mesures des automates. La version définitive en
JAVA est en cours de développement.
Résumé
des travaux: L'objectif est de centraliser le pilotage des différents
automates de culture qui jusqu'à présent ne communiquaient
pas entre eux, de rassembler l'information récoltée et de
la stocker dans une base de données. Enfin, ce logiciel devra permettre
la mise en œuvre des algorithmes de contrôle sur la base des diverses
informations recueillies. La maquette du logiciel a été réalisée,
en TCL/Tk. Les flux d’information et l’interaction entre les divers modules
ont été analysés et modélisés selon
une approche UML. Le logiciel de pilotage est centré sur une base
de données MySQL qui contient les paramètres des divers automates,
et recueille les mesures des automates. La version définitive en
JAVA est en cours de développement.
 Rapport
disponible :
Rapport
disponible :
C.Prévost, O.Croce, O.Bernard et A.Sciandra
 Développement
d'un modèle de croissance du phytoplancton.
Développement
d'un modèle de croissance du phytoplancton.
 Résumé
des travaux: Un modèle, appelé BioLov1, a été
développé. il représente la croissance et la photosynthèse
d'une population de cellules phytoplanctoniques limitées simultanément
par la lumière et l'azote. L'étude mathématique de
son comportement qualitatif montre qu'il transcrit de manière satisfaisante
le comportement entrées-sorties à l’équilibre, c’est
à dire que les tendances observées expérimentalement
en réponse à une augmentation d’un forçage sont respectés.
Des méthodes pour déterminer les paramètres ont été
étudiées et le comportement global du modèle a été
analysé. Le modèle, une fois ses paramètres identifiés
présente une bonne adéquation quantitative avec les mesures
expérimentales. Ce modèle a ensuite été modifié
pour intégrer l’effet de la température. Les réponses
expérimentales qualitatives ont alors permis de valider le comportement
qualitatif du modèle, en réponse aux variations de la température.
Le modèle BioLov2 représente donc l’influence simultanée
de la lumière, de l’azote et de la température.
Résumé
des travaux: Un modèle, appelé BioLov1, a été
développé. il représente la croissance et la photosynthèse
d'une population de cellules phytoplanctoniques limitées simultanément
par la lumière et l'azote. L'étude mathématique de
son comportement qualitatif montre qu'il transcrit de manière satisfaisante
le comportement entrées-sorties à l’équilibre, c’est
à dire que les tendances observées expérimentalement
en réponse à une augmentation d’un forçage sont respectés.
Des méthodes pour déterminer les paramètres ont été
étudiées et le comportement global du modèle a été
analysé. Le modèle, une fois ses paramètres identifiés
présente une bonne adéquation quantitative avec les mesures
expérimentales. Ce modèle a ensuite été modifié
pour intégrer l’effet de la température. Les réponses
expérimentales qualitatives ont alors permis de valider le comportement
qualitatif du modèle, en réponse aux variations de la température.
Le modèle BioLov2 représente donc l’influence simultanée
de la lumière, de l’azote et de la température.
 Rapports
disponibles :
Rapports
disponibles :
L.Pawlowksi, O.Bernard et A.Sciandra
C.Pénard L.Pawlowksi, O.Bernard
et A.Sciandra
 Détermination
des trajectoires à reproduire par le SEMPO
Détermination
des trajectoires à reproduire par le SEMPO
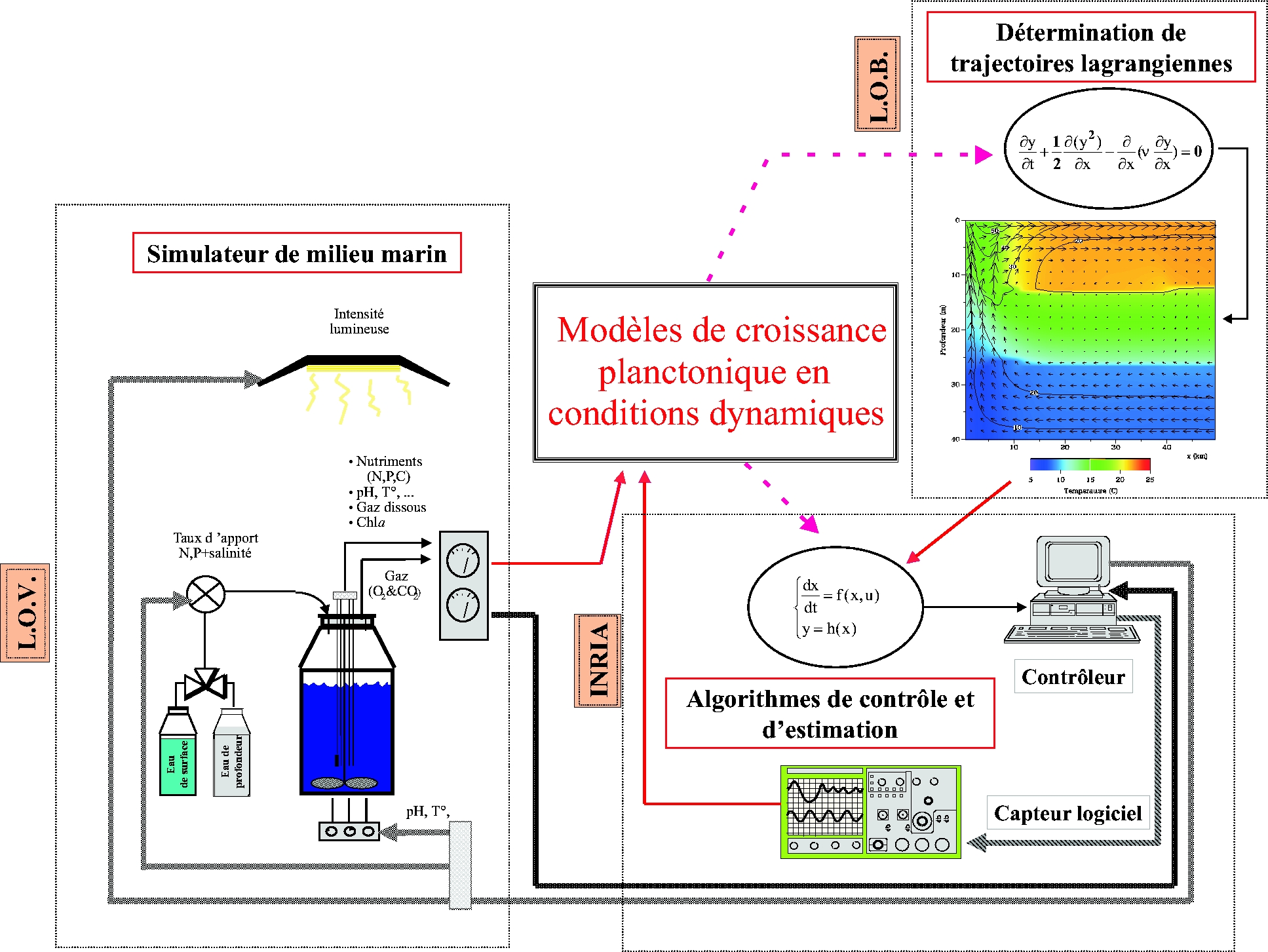
 Résumé
des travaux : Ces travaux s’appuient sur le code de simulation 3-D
d'hydrodynamique côtière SIMPHONIE. Nous avons développé
dans ce code existant un suivi Lagrangien de particules. Le champ de vitesse
calculé par le modèle sert à reconstruire les trajectoires
Lagrangiennes des particules fluides. L'environnement physico-chimique
de la particule peut ainsi être reconstruit. La situation océanique
considérée est schématique : un upwelling côtier
est généré par un vent constant (5 m.s-1).
Nous avons ensuite couplé ce modèle hydodynamique au modèle
BioLov2 décrivant la croissance du phytoplancton en conditions de
limitations par l’azote, la lumière et la température. Les
simulations obtenues ont alors été utilisées pour
reconstruire l’environnement de cellules situées à diverses
positions initiales. Cette étude a ainsi générée
un grand nombre de trajectoires caractérisées par des historiques
physico-chimiques très différentes. Nous avons ensuite procédé
à la classification des trajectoires obtenues afin de faire émerger
des classes de trajectoires. Cette étude montre que 7 groupes peuvent
être considérés. Chaque groupe correspond à
une évolution type de l’environnement d’une cellule, et pourra par
la suite être simulée in vitro par le SEMPO.
Résumé
des travaux : Ces travaux s’appuient sur le code de simulation 3-D
d'hydrodynamique côtière SIMPHONIE. Nous avons développé
dans ce code existant un suivi Lagrangien de particules. Le champ de vitesse
calculé par le modèle sert à reconstruire les trajectoires
Lagrangiennes des particules fluides. L'environnement physico-chimique
de la particule peut ainsi être reconstruit. La situation océanique
considérée est schématique : un upwelling côtier
est généré par un vent constant (5 m.s-1).
Nous avons ensuite couplé ce modèle hydodynamique au modèle
BioLov2 décrivant la croissance du phytoplancton en conditions de
limitations par l’azote, la lumière et la température. Les
simulations obtenues ont alors été utilisées pour
reconstruire l’environnement de cellules situées à diverses
positions initiales. Cette étude a ainsi générée
un grand nombre de trajectoires caractérisées par des historiques
physico-chimiques très différentes. Nous avons ensuite procédé
à la classification des trajectoires obtenues afin de faire émerger
des classes de trajectoires. Cette étude montre que 7 groupes peuvent
être considérés. Chaque groupe correspond à
une évolution type de l’environnement d’une cellule, et pourra par
la suite être simulée in vitro par le SEMPO.
 Rapports
disponibles :
Rapports
disponibles :
P.Cinque et Y.Leredde
G.Mauguin, Y.Leredde, D.Nérini, C.Manté,
O.Bernard, L.Pawlowski et A .Sciandra
Actes de conférences:
Présentations et posters à des
colloques :
-
Pawlowski L., Bernard O., Le Floc'h E., Sciandra A., 2002. Le chémostat:
un outil de développement et de validation de modèles de
croissance phytoplanctonique. Communication présentée
par L.Pawlowski au XXIIème Séminaire de la Société
Francophone de Biologie Théorique - Ecologie Aquatique : Théorie
et Expérience, Saint-Flour, 11 juin 2002.
-
Pawlowski L., 2002. Modélisation de l'effet conjugé
azote/lumière sur la croissance du phytoplancton en chémostat:
étude qualitative et validation. Communication présentée
par L.Pawlowski à l'Ecole de printemps du GDR COREV, ile de Berder,
29 Mai 2002.
-
Pawlowski L., Bernard O., Le Floc'h E., Sciandra A., 2002. Nonlinear
modelling of the coupling between carbon and nitrogen pathway during
phytoplankton growth. Validation with chemostat experiments on Rhodomonas
Salina. Poster présenté par A.Sciandra au 2002 ASLO-AGU Ocean
Sciences Meeting. Honolulu, Hawaii, 11-15 février 2002.
-
Pawlowski L., Bernard O., Le Floc'h E., Sciandra A., 2002. Qualitative
Behaviour of a phytoplankton growth model in a photobioreactor.
Présenté par O.Bernard au congrès IFAC 2002 . Barcelone,
Espagne, 21-26 juillet 2002

![]() Rapport
disponible :
Rapport
disponible :![]() Rapports
disponibles :
Rapports
disponibles :